制限酵素: 反応温度、時間などプロトコールを中心に
UBC/experiments/dna/restriction_enzyme
このページの最終更新日: 2025/11/23- 概要: 制限酵素とは
- 制限酵素反応のプロトコール (single digestion)
- それぞれの制限酵素の特徴
広告
概要: 制限酵素とは
制限酵素 restriction enzyme, restriction endonuclease とは、2 本鎖 DNA 中の特定の認識配列 (recognition sites or recognition sequences) を認識し、切断する 酵素 である (2)。
制限酵素によって DNA が切断される場合、図 (Public domain) のような 2 つのパターンがある。EcoRI のように 5' および 3' 鎖に突出のある末端を

制限酵素 EcoRI による切断パターン |
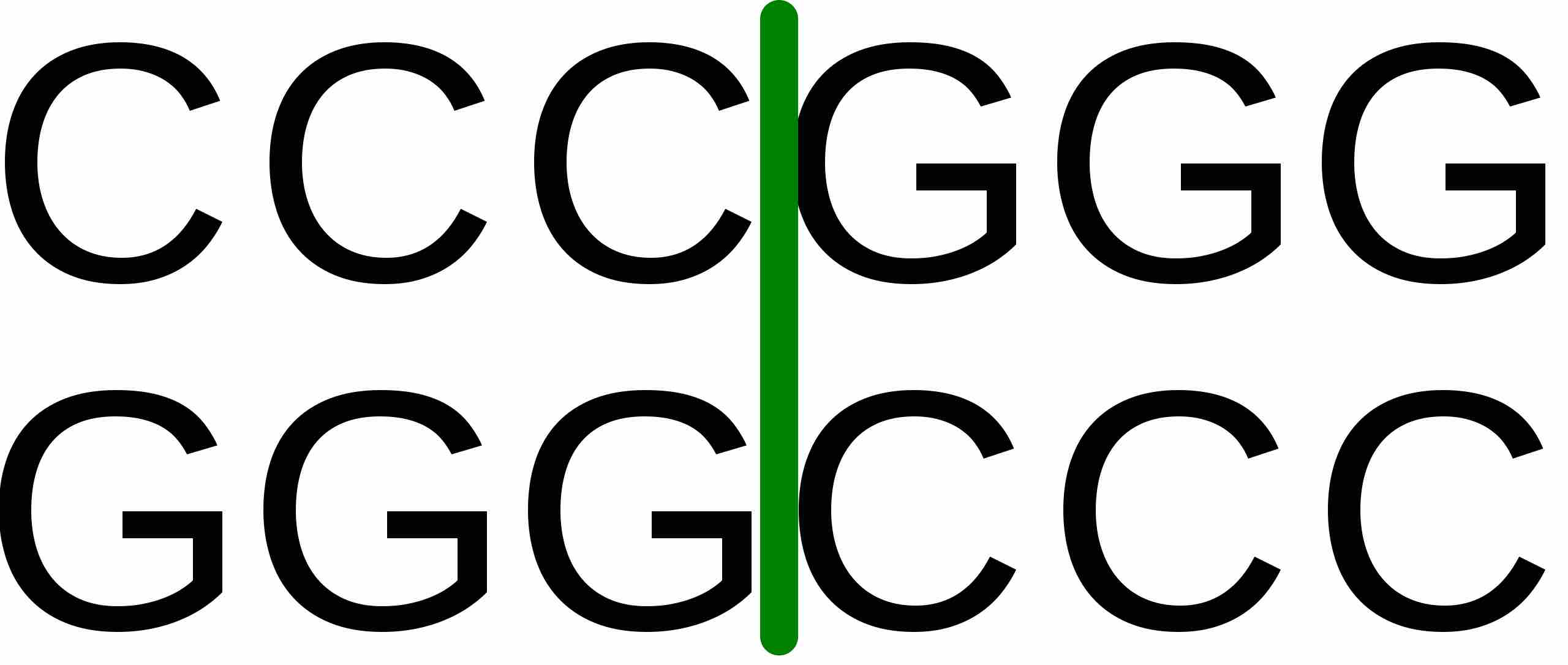
制限酵素 SmaI による切断パターン |
制限酵素は様々な 原核生物 から発見され、本来の機能は外来の (ファージ由来の) DNA を分解することである。制限酵素は、
認識配列の特徴
ほとんどの認識配列は
活性発現機構
通常、Mg2+ などの 2 価イオンが活性発現に必要である (2)。水分子 water からプロトンを引き抜き、DNA のリンを求核攻撃させていると考えられるが、文献 2 には「理由はよくわかっていない」と書かれている。
一つ明らかなことは、
Star 活性
制限酵素は、基質となる DNA に対して大過剰量が存在する場合、特異性が低下して本来の認識配列とは一部異なる塩基配列を切断する場合がある。この現象を制限酵素の
制限酵素反応のプロトコール
もちろん製品によってプロトコールは異なるので、購入した製品のマニュアルを参照してほしい。ここでは、私がよく使っている Takara と NEB の製品を例に解説する。まずは、反応系に 1 種類の制限酵素のみを加える single digestion。
プロトコールは単純で、基本的には DNA、酵素およびバッファーを混ぜるのみで、基本ルールは 2 つ。
- バッファーは全体量の 1/10。
- 酵素は全体量の 1/10 以下にする。Star 活性を抑えるため。
全量が 20 µL のときの反応系は、例えば次のようになる。
DNA 溶液 + 滅菌水 |
17 µL |
バッファー (緩衝液) |
2 µL |
制限酵素 |
1 µL |
合計 |
20 µL |
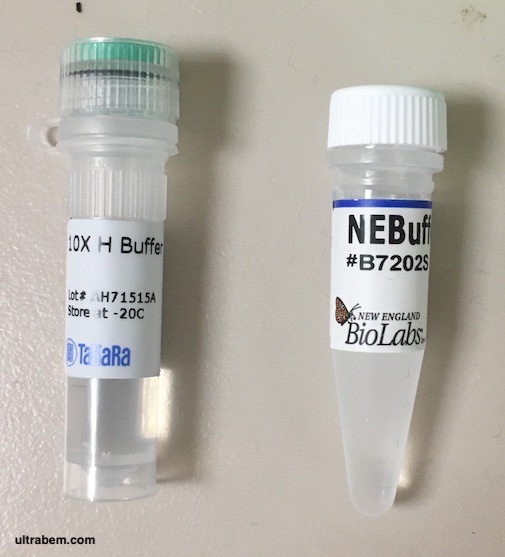
Takara、NEB 制限酵素緩衝液の選択と組成
制限酵素は、緩衝液の種類によって活性が異なる。Takara では、基本的にキャップの色で合わせれば良いことになっている。下の写真 (Public domain) で、10 x H と書かれている緑のキャップのチューブが Takara の H バッファーである。つまり、緑色の蓋の制限酵素を使うときには、この 10 x H バッファーを使えば良い。
NEB のバッファーは、NEBuffer という名前がついており、さらに 1, 2, 3.. のように番号がつけられている。チューブに色はついていないので、カタログを参考に使うバッファーを決定する。
組成も上記のリンク先に載っている。基本的には Tris, MgCl2, DTT, NaCl, KCl などで、特に変わった試薬は使われていない。

|
|
酵素量・反応時間をどう決めるのか
制限酵素反応液の組成を決める際には、
たとえば、タカラの制限酵素活性の 1 U (ユニット) は、「酵素反応液 50 µL 中、37 ℃、1 時間に 1 µg の λ DNA を完全に分解する酵素量」と定義されている。制限酵素溶液の活性は製品によって異なり、「10 U/µL」などのように記載がある。
したがって、10 U/µL の酵素を 1/10 含む反応液の場合、最終的な酵素量は 1 U/µL となる。DNA が 1 µg ならば、1 時間の反応で十分であると考えることができる。ただし、DNA の高次構造などによって厳密なところは変わってくるはずなので、ここから少し長めにとるのを標準プロトコールとしていることも多いだろう。
ベクター構築後のインサートの確認など多少の切れ残りがあっても良い場合、ligation 前など切れ残りを極力減らしたい場合など、状況に応じて判断する。
制限酵素・個別の特徴
プライマー設計などで、細かくチェックしたことがあるもののみ。
BamHI |
5'- G|G A T C C -3' | |
BglII |
5'- A|G A T C T -3' | |
DpnI |
5'- G A*| T C -3' | A* はメチル化塩基。Inverse PCR で使われる。 |
EcoRI |
5'- G|A A T T C -3' | |
KpnI |
5'- G G T A C|C -3' | |
NdeI |
5'- C A|T A T G -3' |
広告
References
- 制限酵素の活性の定義、純度検定、保存、添付Bufferについて. Takara Website. Link.
- Amazon link: ストライヤー生化学
: 使っているのは英語の 6 版ですが、日本語の 7 版を紹介しています。参考書のページ にレビューがあります。
- 分子生物学実験の基礎:制限酵素処理. Link: Last access 2020/08/20.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
