PCR プライマー: 設計方法、汎用プライマーの配列など
UBC/experiments/dna/pcr_primer
このページの最終更新日: 2025/11/23- プライマー設計で重要なパラメーターと基本ルール
- 長さ
- GC 含量
- TM 値
- 二次構造
- 特異性
- プライマー設計用のウェブサイト
- ロング PCR 用のプライマー
- 縮重プライマー
- 汎用プライマーの配列
別ページ
広告
プライマー設計で重要なパラメーターと基本ルール
プライマー設計では、多くのパラメーターが「あちらを立てればこちらは立たず」という状況にある。したがって「常にベストなプライマー配列」というものは存在せず、PCR の目的に応じて最適なプライマーを設計する必要がある。考慮すべきポイントの概要は以下の通り。
このページでは、長さ、GC 含量などの項目についてまず一般的な内容を述べ、次に リアルタイム PCR、ロング PCR などのアプリケーションについての情報がある場合には、それらを追記するという形をとる。
長さ
18 - 30 塩基程度。長いプライマーは特異性が上がるが、アニーリング効率が下がるので収量は低下する (1)。
GC 含量
- 40-60% にする (1)。
- G および C が連続して存在しないようにする (1)。
Tm 値
- 55-65°C にする (1)。
二次構造
プライマーが二次構造を作らないような配列にする (1)。
特異性
プライマー配列を BLAST したときに、forward と reverse が同じ遺伝子に top hit するか? 塩基同一率と e-value は?
プライマー設計用のウェブサイト
様々なウェブサイトがあるが、管理人が現在使っているのは Primer-BLAST で設計し、その配列を IDT Oligo Analyzer でさらに検証するという方法である。ただしこれはリアルタイム PCR プライマーの設計時で、普通の PCR プライマーのときは IDT Oligo Analyzer によるチェックは省くことが多い。
Crafting qPCR Primers: NCBI Primer BLAST for Beginners という論文 の流れに極めて近い。
Primer-BLAST
2024 年上半期、Google Scholar で 2270 件の論文がヒットし、下記の Primer3 よりも使われている様子。BLAST と組み合わさっているので、生物種を指定すると、他のテンプレートから増えてしまう可能性のあるペアには Products on potentially unintended templatesという警告が出る。
Primer3
かなり昔からあるソフトだが、Google Scholar で Primer3 + qPCR で検索したところ、2024 年上半期に出版されたものだけでも 1650 件の論文がヒットした。まだ十分に使われているようだ。
Google 検索の結果で目立つのは Primer3 だが、Primer3plus の方がいろいろと便利。プライマーをピックアップした後、Primer 3 Manager を使って BLAST したりすることができる。
IDT Oligo Analyzer
IDT が提供するウェブページ。IDT のアカウントが必要。
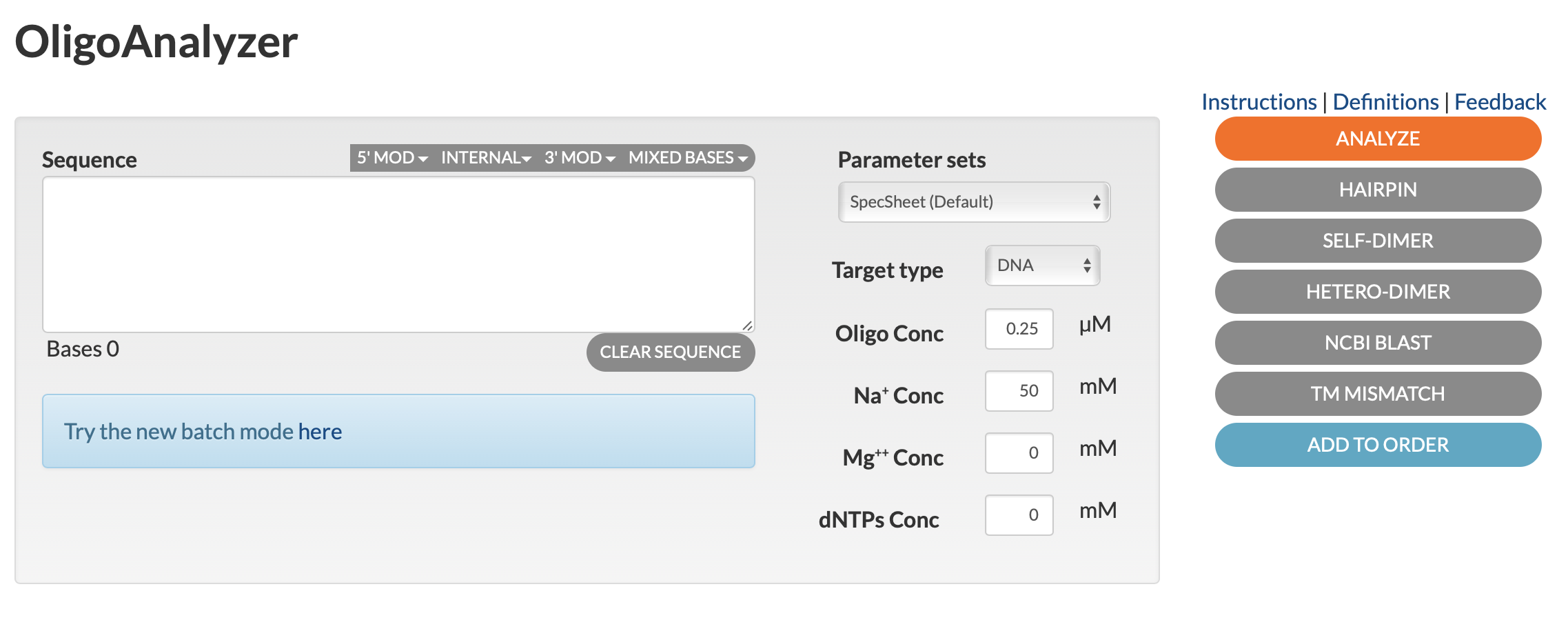
使用方法は以下の通り。
- プライマー配列をボックスに入力し、各種パラメーターを設定する。
- Oligo conc は通常 0.25 µM 程度。デフォルトでこの値に設定されている。
- Mg2+ 濃度は、一般に 1 - 4 mM、dNTP 濃度は一般に 0.2 mM each。
ヘアピン構造の予測では、Gibbs の自由エネルギー変化などの熱力学的パラメーターまでちゃんと出てくる。

ΔG の単位は kcal/mol など。ヘアピン構造を取る際の自由エネルギー変化。この値がマイナスであれば、ヘアピン構造の方がエネルギー状態が低いということで、この反応が自発的に起こる。つまり、放っておくとプライマーがどんどんヘアピン構造に変化していくということ。
上の例の場合、自由エネルギー変化は正であるので、この反応は自発的には起こらない。ただし、自由エネルギー変化の値が小さいということは、その構造変化が起こりやすいということでもある。
PCR の場合、ヘアピン形成の際の自由エネルギー変化だけではなく、プライマーとテンプレートが二本鎖を形成する際の自由エネルギー変化も重要な因子である。両者を混同しないように注意すること。
プライマーダイマーの予測は、homo(self)-dimer と hetero-dimer に分かれている。もちろん、hetero-dimer 形成を予測したい場合は、もう一つのプライマーの配列を入れる必要がある。この場合も、自由エネルギー変化 ΔG が計算される。

DNA は基本的に二本鎖になっていた方が安定なので、この場合、ΔG は基本的に負になる。負の絶対値が大きいほど、プライマーダイマーが形成されやすくなる。どの程度なら OK かというのは場合によるが、一般的な指標としては、ΔG は
Crafting qPCR Primers: NCBI Primer BLAST for Beginners に、ヘアピンとΔGに関する言及がある。
- Ensure that the melting temperature of the hairpin structures is lower than the annealing temperature of the PCR reaction (50°C).
- Make sure the ΔG of the homodimers are greater than -9 kcal/mole (so, less negative values or positive values).
その他ソフト
Beacon Designer というのもあって、これは有料。ただしフリートライアルも可能。
PrimerExpress は旧 ABI の時代からあったソフト。これも有料。
ロング PCR 用のプライマー
- 長いプライマーを使う。文献 2 では 26 塩基。
- Tm 値を高めにする。文献 2 では 68°C。
プライマー配列がユニークであること、標的配列中に SNP がないこと。これらは普通の PCR プライマーでも重要であるが、より難しいロング PCR ではクリティカルである。
縮重プライマー
多くの生物でゲノム配列が手に入る現在、縮重プライマーに対する需要は激減している。CODEHOP や HYDEN のように有名だった縮重プライマー設計ツールも閉鎖しているという状態だ。
PubMed からのリンクが働かない。 |
|
Last update 2008。Windows 用のコマンドラインソフトをダウンロードできる。 |
|
CODEHOP |
|
Java ベースのソフトウェアなので、Java 環境をインストールする必要がある。 |
DECIPHER の primer 設計は、複数の配列から target 配列を特異的に増幅するプライマーを設計するためのもので、縮重プライマーとは別のものだった。2019 年 2 月 11 日に修正。
2018 年 8 月に探してみたところ、この DECIPHER というサイトがオンラインの縮重プライマー設計ツールとして唯一使えそうなものだった。これを見つけるのにも 30 分ぐらいかかった。
使用方法はけっこうクセがある。
- FASTA file をアップロードするが、aligned fasta という珍しい形式を要求する。Clustal Omega でアウトプットを fasta に指定し、結果をダウンロードすれば得ることができる。
- FASTA file に含まれる配列のうち、一つをターゲットに指定する必要がある。ターゲットは > 以外の部分の配列名で、スペースなどがあるとうまく認識されなかった。
- 私の場合、結果はブラウザ上に表示されず、Detailed なんとかというところをクリックするとコンマ区切りテキストとして現れる。
縮重塩基の一文字表記
ThermoFisher のプライマー注文画面から見ることができる、塩基の IUPAC code をまとめておく。通常の A, T, C, G, U の他の塩基である。
塩基 |
IUPAC Code |
Deoxyinosine |
I |
A + C + G |
V |
A + T + G |
D |
T + C + G |
B |
A + T + C |
H |
A + T |
W |
C + G |
S |
T + G |
K |
A + C |
M |
C + T |
Y |
A + G |
R |
A + G + C + T |
N |
汎用プライマーの配列
汎用プライマーの配列は、文献によって多少配列が異なる場合が多い。出典とともに示しておく。
| 名称 | 配列など (5' - 3') |
M13F |
|
M13F43 |
|
M13R |
|
M13R49 |
|
T7 promoter |
|
T7 terminator |
|
T3 promoter |
|
sp6 promoter |
|
CMV forward |
|
References
- Roche オンラインマニュアル. プライマー設計とテンプレートの調製. Pdf file.
- 国立遺伝学研究所 細道一善. 疾患関連遺伝子の long-range PCR Nextera 解析. Pdf file.
広告
アップデート前、このページには以下のようなコメントを頂いていました。ありがとうございました。これはおそらく、プライマーが長いと Tm 値も上がるため、同じ温度でのアニーリング効率が低下するということだと思います。
Tm 値にはいくつか計算方法がありますが、たとえば このページ に例が載っています。基本的には、プライマーが長いほど、また GC 含量が高いほど Tm も高くなります。
|
2018/10/20 20:35 長いプライマーを用いた際、アニーリング効率が低下するのはなぜでしょうか? |
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
