インバースPCR: プライマー設計、プロトコールなど
UBC/experiments/dna/pcr_inverse
このページの最終更新日: 2025/11/23広告
概要: インバース PCR とは
インバース PCR (inverse PCR) とは、プライマーを設計した既知配列に隣接する領域を増幅する PCR である。以下のように、環状 DNA を鋳型とする。
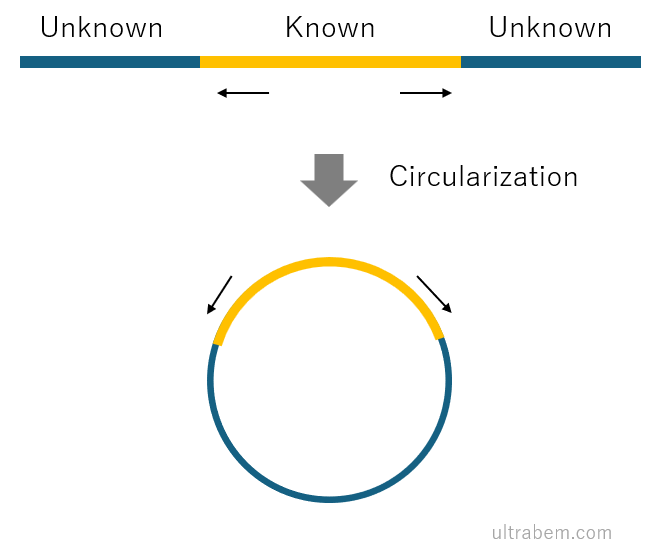
もともとは、長いゲノム DNA 断片などを読み進めていくために開発された手法だと思われる。現在では、タンパク質発現系でベクターを改変したり、遺伝子に変異を導入したりするために使われることが多いだろう。
Inverse PCR を行う際の、一般的な注意点。
- プライマーは、アニール部分を 25 塩基以上 (1)。
- 正確性の高い酵素を使う。KOD-Plus-Neo などの使用例がある。
インバース PCR を用いた変異導入プロトコール
一般的な手順は以下の通り。
- プライマーの設計
- Inverse PCR
- DpnI 処理による鋳型プラスミドの分解
- セルフライゲーション
- 大腸菌の形質転換
プライマーの設計
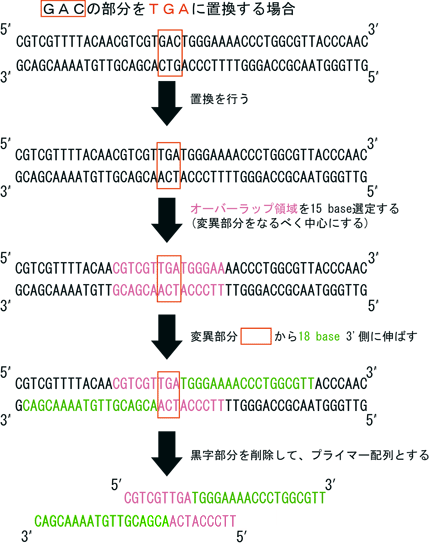
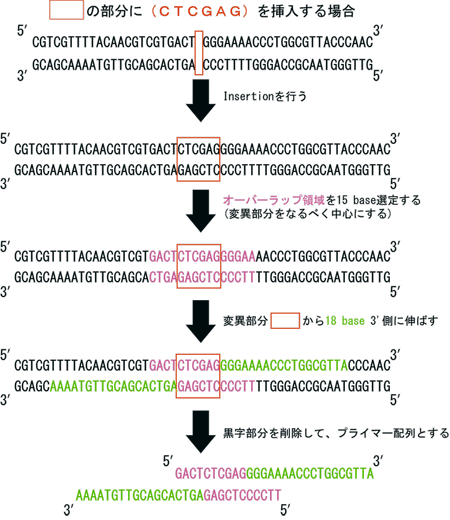
Takara のページ に非常にわかりやすい図があったので引用する。
変異の部分を中心に 15 塩基を選定、そこから左右に 18 塩基伸ばして設計するのが基本のようだ。
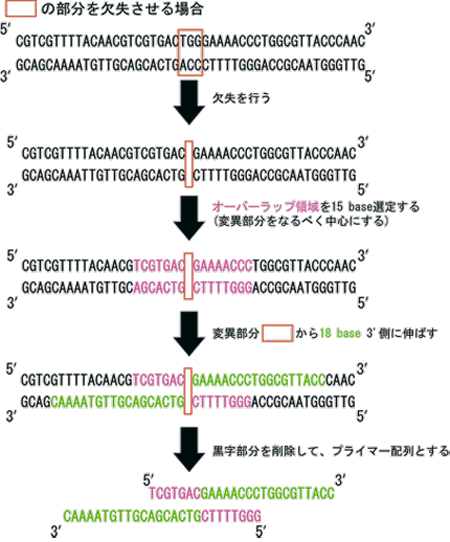
置換および挿入の場合も、原理は同じである。
プライマーのリン酸化
通常のベクターへのライゲーションと違い、リン酸化が問題となる。リン酸化の詳細は ライゲーションのページ も参照のこと。
通常のライゲーションではベクターがリン酸化されているが、inverse PCR で増やした DNA 断片はリン酸化されていないので、そのままではセルフライゲーションすることができない。したがって、5' 側にリン酸基を付加する必要がある。これにはいくつか方法がある。一般に使われる酵素は T4 polynucleotide kinase (T4PNK) である。
リン酸基を付加するために ATP が必要であるが、これは T4PNK のバッファーに入っている。説明書を見ると、リン酸基の付加効率を調べるために放射性の ATP を使うプロトコールが載っていたりして少しびびるが、通常の ATP で良い。
T4PNK のキットを用いて、プライマーをリン酸化する。
Inverse PCR
文献 2 によると PCR は 4 - 10 サイクルである。その前に、余計なバンドが出ないことを アガロースゲル電気泳動 で確認する予備実験として 10 - 20 サイクルの PCR をすると書かれている。セルフライゲーション後に形質転換するので、電気泳動ではっきり見えるほど増やす必要はない、ということ。
また、PCR → DpnI 処理 → セルフライゲーションの過程で、DNA のカラム精製は行わない。原理的には、DpnI で断片化されたプラスミドが挿入されてしまうはずだが、その効率はセルフライゲーションに比べて著しく低いようだ。
DpnI 処理による鋳型プラスミドの分解
DpnI は、メチル化された DNA を特異的に分解する珍しい 制限酵素 である。認識配列は以下の通りで、A* がメチル化 アデニン。したがって、
5'- G A*| T C -3'
3'- C T | A* G -5'
これによって、のちのトランスフォーメーションで、新しく作られたプラスミドだけが使われることになる。
セルフライゲーション
DpnI 処理したプラスミドをセルフライゲーションする。
プライマーのリン酸化をしていない場合は、この時点で T4PNK 処理をすることも可能である。DNA ligase のバッファーに ATP が入っているので、ライゲーション溶液に T4PNK 溶液を入れておくプロトコールが一番簡単である。ただし、上にも書いたように、リン酸化反応は一本鎖 DNA の方が効率がいいので、プライマーをリン酸化しておくのが一番よいように思う。
広告
References
- インバースPCRを用いた部位選択的な遺伝子変異の導入. Link: Last access 2025/03/05.
- KOD -Plus- Mutagenesis Kitを用いる部位特異的変異導入. 東洋紡プロトコール (Pdf): Last access 2025/03/06.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
