IPTG によるタンパク質の発現誘導: 原理、プロトコールなど
UBC/experiments/protein/recombinant_ecoli_iptg
このページの最終更新日: 2025/11/23関連ページ
- ベクター関連の用語集: タグの一覧など
- この実験を行うのに必要な手続き
- 試薬のページ: IPTG
広告
概要: IPTG による発現誘導の原理 (pET システム)
IPTG による発現誘導は、大腸菌を使ったタンパク質発現系でおそらく最も頻繁に使われる方法である。ラクトースオペロン利用した発現システムである。ラクトース のページに、ラクトースオペロンに関する記述がある。
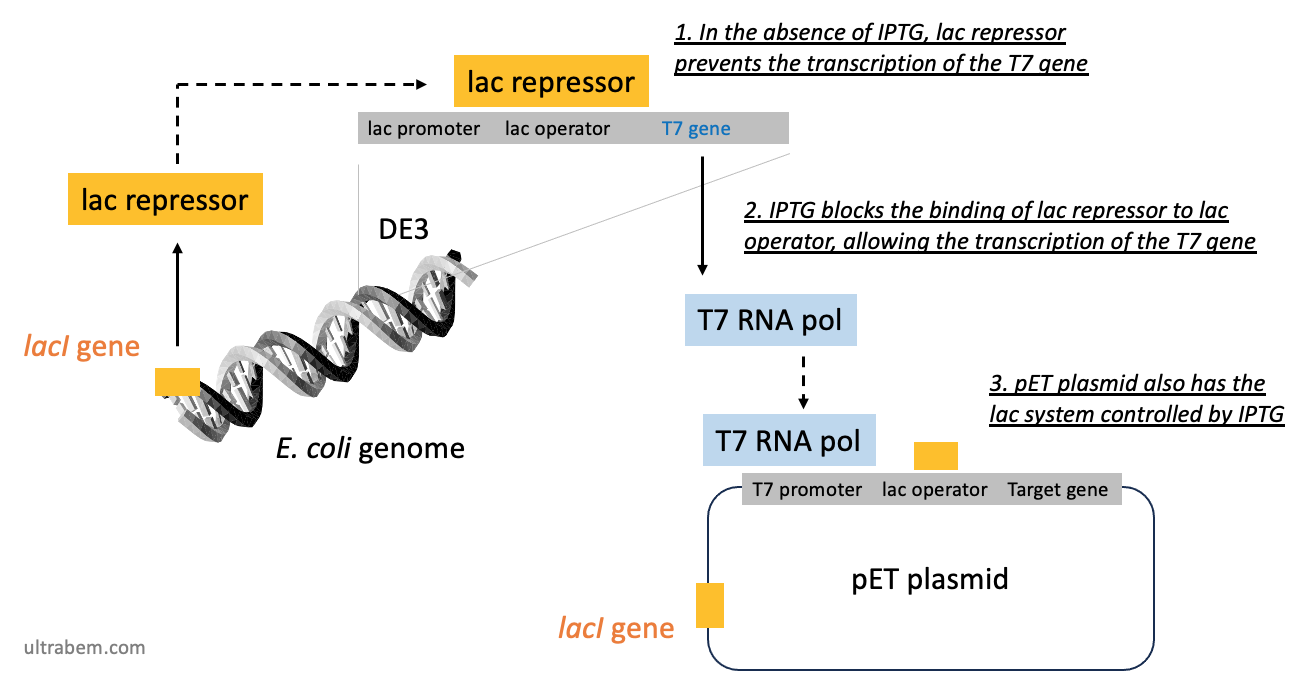
ここでは、汎用される pET ベクターを使った方法の原理について述べる (1)。pET システムでは、IPTG が直接ベクター上のプロモーターに作用するのではなく、大腸菌の T7 RNA polymerase を介して発現を誘導する。
- 大腸菌ゲノムにある lacI 遺伝子から、lac リプレッサーが発現する。ラクトースまたはその類縁体がないとき、lac リプレッサー は DE3 という領域の lac operator に結合し、T7 RNA ポリメラーゼの転写を抑制している。
- したがって、pET 発現システムでは L8-UV5 lac プロモーターの支配下に T7 RNA ポリメラーゼがある大腸菌を使わなければならない。
- この L8-UV5 lac プロモーターという構造は、λDE3 というバクテリオファージに含まれる。
- したがって、pET システムに使える大腸菌には、このバクテリオファージがゲノム中に含まれていなければならない (溶原サイクル lysogenic cycle)。たとえば DE3 を含む BL21 株であり、これは BL21(DE3) のように表される。
- IPTG を添加すると lac リプレッサーが解離し、T7 RNA ポリメラーゼが転写されるようになる。
- この T7 ポリメラーゼは、pET プラスミドの T7 プロモーターに結合し、下流に組み込まれた遺伝子を転写する。pET plasmid にも lacI 遺伝子と lac operator があり、IPTG 非存在下での転写を二重に防ぐ形になっている。
参考に、ラクトースオペロンの概略図も載せておく。
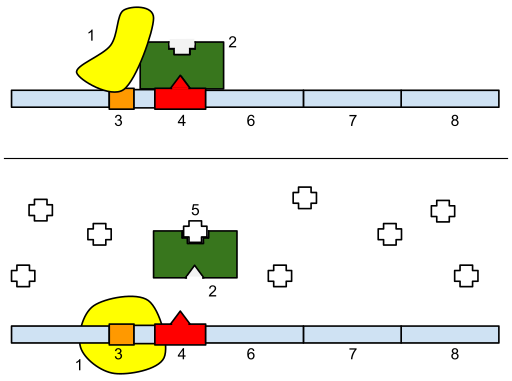
ラクトースオペロンの概略図 (2)。上は転写が抑制されている状態、下はラクトースや IPTG によって転写が活性化された状態。1. RNA polymerase、2. lac リプレッサー、3. プロモーター、4. オペレーター、5. ラクトース (IPTG)、6. lacZ、7. lacY、8. lacA. |
- 大腸菌の T7 RNA ポリメラーゼが、pET ベクター上の T7 promoter に作用し、ベクターに組み込まれた遺伝子を転写する。
L8-UV5 lac プロモーター
L8-UV5 lac プロモーターとは、lac プロモーターに L8 と UV5 という 2 つの変異が導入されたものである (1)。
lac プロモーターは、培地中の グルコース 濃度が低下すると活性化する。L8 変異は、この活性化を抑制している。このため、培養条件によらず T7 RNA ポリメラーゼの転写量が低く保たれる。
UV5 変異は、文献 1 では「L8 変異のサプレッサー」と書かれている。おそらく、IPTG 添加時に、L8 変異の影響を抑制し、T7 RNA ポリメラーゼが多く作られるようになっている。
なお、T7 RNA ポリメラーゼの特異性は非常に高く、T7 プロモーターにしか作用しない (1)。
カタボライト抑制
ラクトースが存在していても、グルコースがある場合にはそちらが優先的に使用されるメカニズムがある。これをグルコース効果またはカタボライト抑制という (参考ページ)。
カタボライト抑制のメカニズムは、概ね以下の通り。
- cAMP-activated protein CAP はアクチベーターであり、Lac operon の発現を正に制御している。
- グルコースは細胞内 cAMP 濃度を下げ、これによって CAP が抑制される。
- したがって、グルコース存在下では Lac operon の活性は低く抑えられている。グルコースがなく、ラクトースが存在するときに Lac operon の活性は相加的に上昇する。
プロトコール
IPTG ストック溶液の調製
一般に 0.1 M IPTG をストック溶液として用いる。調製方法は以下の通り。
- IPTG の 分子量 は 238.3 なので、
0.238 g を 10 mL の滅菌水に溶解 する (5)。 - 溶液はろ過滅菌し、1 mL ずつ分注して -20 °C に保存。
なお、LB 培地に IPTG を加えるときは、LB 培地のページ にあるように終濃度 0.2 mM 前後で加える。1 M をストック溶液にしているラボもあるようだ。
IPTG は、β-ガラクトシダーゼに分解されないため、一度添加すると培地中の濃度が一定に保たれるというメリットがある。ただし、natural sugar に比べて菌に対するストレスになるという論文もある (6I)。コストの低減を目的に、IPTG でなく skim milk の使用を検討している論文もあった (6)。
発現誘導の条件検討
大腸菌発現系では、最大量の発現タンパク質を可溶性画分として得ることを目指し、発現誘導プロトコールを最適化する必要がある。主に検討する誘導条件は以下の 4 つである。
個人的には、文献 4 の初期設定と検討範囲が極めて妥当であると思う。
発現誘導時の密度 |
大腸菌の密度は 600 nm の吸光度 (OD600) の値で表す。 0.4 を試してから、0.2 - 0.6 の間で最適化 (4)。 ただしこれは実験系により、1 - 2 ぐらいで最もよくワークする場合もある。 |
培養温度 |
37°C を試してから、18 - 30°C の間で最適化 (4)。 培養温度が高いと、発現タンパク質が封入体に行ってしまう可能性が上がる。これを防ぐために pCold などの低温誘導性ベクターも販売されている。 |
IPTG の濃度 |
0.4 mM を試してから、0.1 - 0.6 mM で最適化 (4)。 |
発現誘導後の培養時間 |
2 時間を試してから、1.5 - 4 時間で最適化 (4)。 |
T7 プロモーターはかなり強力なプロモーターなので、IPTG による発現誘導は原則として大腸菌に対してストレスとして働く。大腸菌が十分に増えてから誘導をかける手法もよくつかわれている。これはプレート培地でも可能で、たとえば大腸菌をニトロセルロース膜の上にまいて、コロニーが形成されたら、膜を IPTG を含む培地に移せば OK。
発現タンパク質の抽出
培養液に IPTG を添加して発現誘導をかけると、mRNA およびタンパク質の発現が誘導されるはずである。たとえば この論文 では、遺伝子 4 個が一つのプラスミドに乗っている系で、それぞれの遺伝子の mRNA 量が 8 倍程度まで増大。ただし、この論文は最適な 内部標準遺伝子 を探すことが目的の論文で、内部標準によってこの数値が変わるというのがメイン。
タンパク質の抽出 のページも参照のこと。ポイントになるのは以下の点である。
- 発現タンパク質が、大腸菌の細胞質画分に可溶性タンパク質として存在するのか、封入体に入っているのか。
- どんな方法で、発現タンパク質の分解を防ぎつつ、効率的に細胞を破砕するのか。
発現タンパク質の精製
タグの種類によって異なる。
His tag をつけて発現させた場合、Ni レジンなどを買って精製することが多いと思う。その場合は、キットに溶出バッファーが含まれている。
どんなデータを論文に載せればいいのか
大腸菌で作ったタンパク質を使った実験では、形質転換、発現、抽出、精製などの過程でたくさんのデータが得られる。通常、もっとも興味があるのは発現タンパクの機能 (結合、酵素活性など) であり、そこに至るまでの予備的データをどの程度論文に載せればいいのか迷うところである。
そこで、大腸菌発現系を扱っているオープンアクセス論文から、いくつか図を拾ってみた。
まずは Dobson et al. (2001) である。Legend とともに引用する。レーン 5 から 7 は切り貼りであり、ここでははっきりとわかるから良いものの、本来は別のゲルであることを明示する方がよい。
この図のポイントは以下の通り。
- タイトルは Bacterial expression of recombinant *** である。PfPP5 はもちろんタンパク質名。
- His-tag だが、Western blot ではなく、IPTG 誘導前、誘導後、精製後の SDS-PAGE を載せている。WB は文章で data not shown として言及されている。
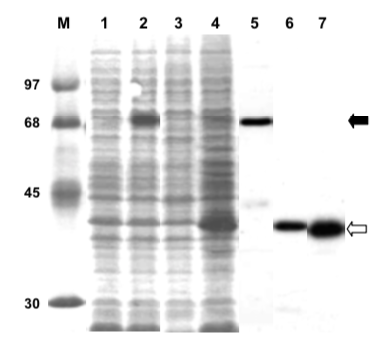
Bacterial expression of recombinant PfPP5. Soluble S100 extract (approximately 30 μg protein) of IPTG-induced (lanes 2, 4) or uninduced (lanes 1, 3) E. coli BL21(DE3)-RIG containing pET-15b-PfPP5 (lanes 1, 2) or pET-15b-ΔTPR-PfPP5 (lanes 3, 4) were analyzed by SDS-PAGE followed by staining of the gel with Coomassie Brilliant Blue R250 [12]. The respective purified proteins were analyzed in lanes 5, 6. Lane 7 represents about 5 μg of purified His-tagged PfPP5 digested with 50 ng of trypsin for 30 min at 30°. Protein markers (lane M) are indicated by their Mr in thousands.
別のパターンも見てみよう。Pennati et al. (2014) では、図のように IPTG 添加後の経過時間のデータがある。レーン 1, 2, 3 がそれぞれ IPTG 添加後 1, 3, 16 時間。A は SDS-PAGE, B は WB。このほか、続く図に 精製過程の elution pattern と SDS-PAGE がある。
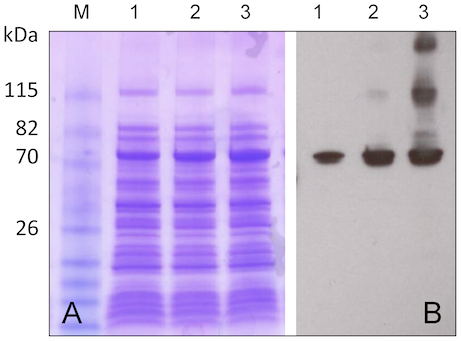
とりあえずの結論として、IPTG なしのネガコン、IPTG 誘導後のサンプルについて精製前と精製後の SDS-PAGE、さらに精製後の WB があれば問題ないと言えそうだ。IPTG 添加後の時間経過はオプションで良いのではないかと思う。
広告
References
東端 2013a (Review). 大腸菌を宿主とした異種タンパク質高発現のイロハ. 生物工学 91, 96-100.- By T A RAJU - 投稿者自身による作品, CC 表示-継承 3.0, Link
- 25℃でIPTGによるタンパク発現誘導をする方法. Link: Last access 11-9-2017.
- Lablogue 大腸菌を用いたタンパク質の発現 4 Link: Last access 2017/11/09.
- IPTGの調製、使用法. タカラバイオ. Link: Last access 2019/01/18.
Khani & Bagheri, 2020a. Skimmed milk as an alternative for IPTG in induction of recombinant protein expression. Protein Expr Purif 170, 105593.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
