In-fusion クローニングによるベクター構築:
原理、プロトコール、プライマー設計など
UBC/experiments/dna/vector_in_fusion
このページの最終更新日: 2025/11/23広告
概要: in-fusion とは
In fusion とは、PCR 断片と線状化ベクターの末端にある 15 塩基の相同配列を利用して、効率よくクローニングする技術である。
- 相同部分が 12 塩基以下または 21 塩基以上だと、in-fusion の効率が低下する。プライマーに付加する配列は 15 塩基、複数断片の in-fusion の場合は 20 塩基が推奨。
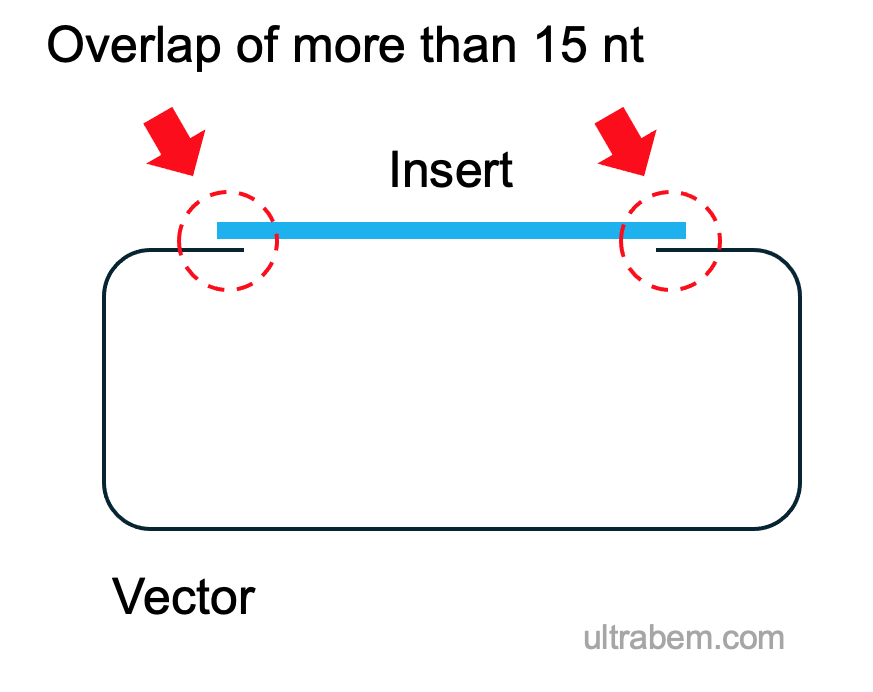
In-fusion プライマーの設計
たとえば、マウス mouse の メラノコルチン-3受容体 遺伝子の翻訳領域を、in-fusion を使って pUC19 というベクターに組み込みたいとしよう。PubMed で検索対象を Nucleotide にし、mouse melanocortin で検索すると、melanocortin 3 receptor (Mc3r), mRNA (NM_008561.3) がみつかる。塩基配列は以下の通り。下線部分が翻訳領域である。
まず、タカラバイオのページ にある Design your primers から、デザインツールを launch する。
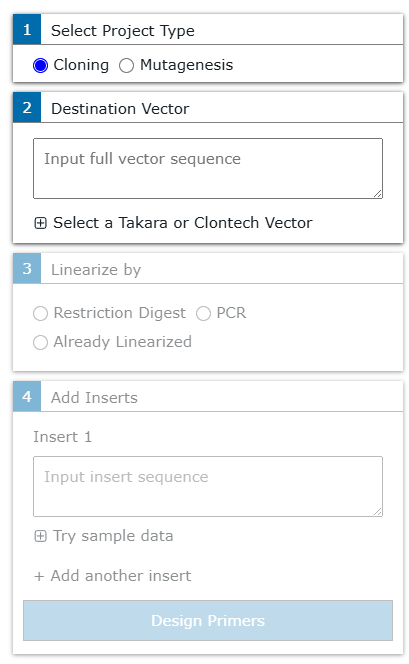
入れるべき情報は 4 種類。まず、今回の目的は Mutagenesis でなく Cloning。次に、ベクター配列の全長。今回、例として使う pUC19 の配列は以下。全長をコピペして入れよう。
In-fusion では、ベクターは何らかの方法で直鎖化されていなければならない。方法として、制限酵素処理、PCR、または「既に直鎖化されている」が選べる。いずれの場合にも、直鎖状になっている端っこの配列が重要なので、ベクターがどういう状態にあるかは正確に示さなければならない。
「制限酵素処理」を選ぶと、このようにベクターマップが自動で表示される。自動的に Single cutter、つまり切断箇所が一個のみの制限酵素が選ばれる。切断箇所が複数あると、ベクターが細切れになってしまうので、これは当然である。
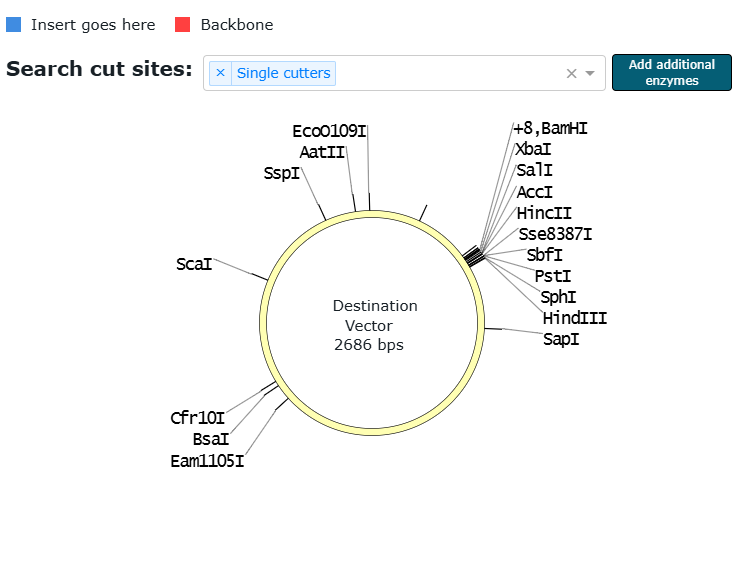
通常は、たくさん制限酵素認識配列が並んだマルチクローニングサイトに遺伝子を入れるだろう。制限酵素の一つをクリックすると、このようにカットの詳細が出てくる。
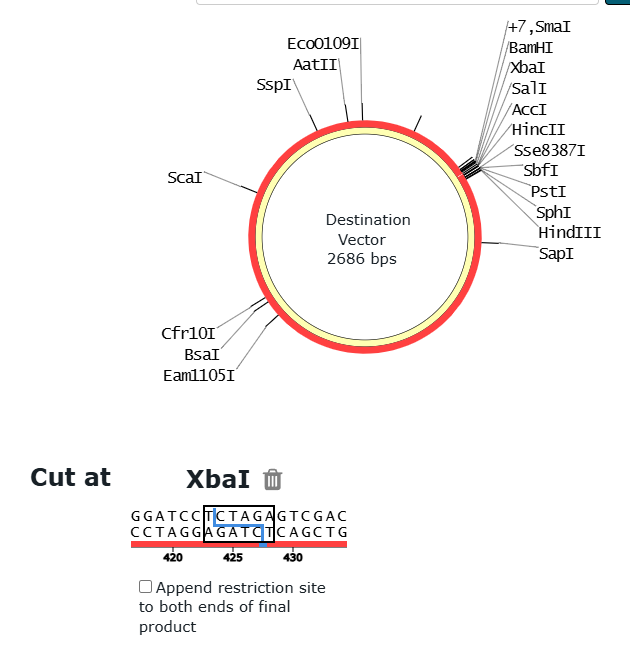
最後に、インサートの配列を入れる。とりあえずは単一断片の in-fusion ということで、インサートは一種類。上記のマウス・メラノコルチン-3受容体の翻訳領域 (下線部分) をコピペして、Desing Primers をクリック。
結果は以下のように表示される。まずはベクターマップ部分。ベクターとインサートの境界が、塩基配列レベルで表示される。
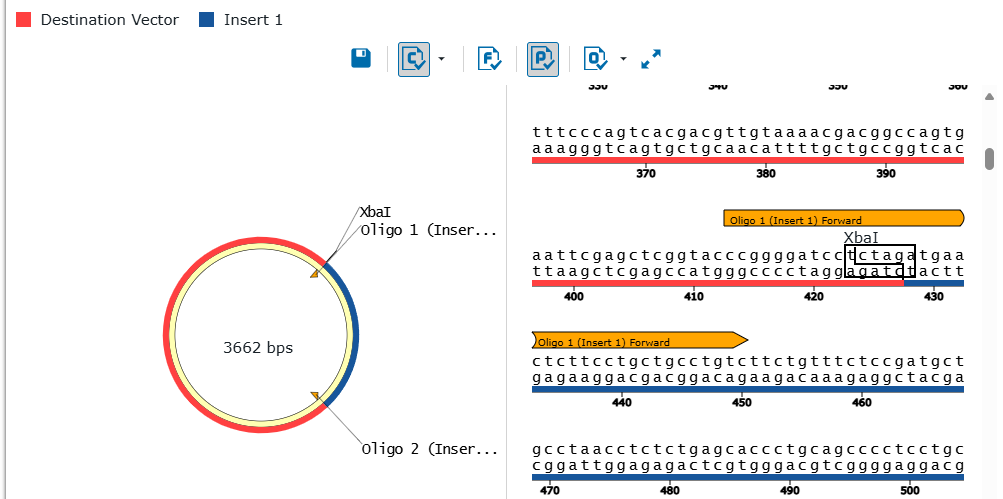
In-fusion は、ハングオーバーがあっても組み込める。制限酵素で切った部分はこのようになっていて、突出末端の方を数えて 15 塩基のオーバーラップがあればいい。

その下に、プライマー配列が表示される。Tm 値などの情報も出てきて、そのままコピペして注文すればよい形になっている。
In-fusion メモ
制限酵素サイト付きのプライマー で増幅した DNA をベクターに ライゲーション するのが昔のスタンダードであったが、いまは in-fusion の方がはるかに効率がいい。面白いのは、場合によっては制限酵素サイト付きのプライマーで増やす必要はまだあるということ。つまり、まずベクターを 2 種の制限酵素でカットする。次にそれらの制限酵素サイトをもつプライマーをこのページで書かれているように設計するが、これは制限酵素処理する必要はない。相同な配列があるので、in-fusion でそのまま入る。ベクターのみが処理されていてもクローニングできるのが in-fusion の強みである。
ベクター + 2 断片の in-fusion は比較的簡単で、融合タンパク質を作ったりするのに有効。ベクター + 3 断片は、濃度をしっかり揃えたりしないと難しく、効率がかなり落ちる印象である。
大きい断片のクローニング。Takara のページ によると、15 kb までは十分な効率でクローニング可能と書いてあるが、経験上はやはり大きくなるほど効率は落ちる。インサートの高次構造など、いろいろな要素があるのでケースバイケースだろう。一断片ずつ入れると、少しは効率の悪さをレスキュー出来る。
広告
References
- Snap Gene pUC 19 vector. Link.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
