分岐鎖アミノ酸合成の最初の酵素
アセト乳酸合成酵素 (ALS): 構造、機能など
protein_gene/a/als
2018/06/11 更新
- 概要: ALS とは
- アイソフォーム
- 構造
- 酵素としての特徴
- Regulatory subunit と活性
- BCAA によるフィードバック阻害
- ALS の阻害剤 (別ページ)
広告
概要: ALS とは
以下の図は Val 生合成 の第一段階で、2 分子の ピルビン酸 からアセト乳酸および 二酸化炭素 を合成している。
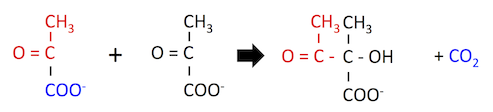
ALS は、1 分子のピルビン酸と 1 分子の 2-ketobutyrate を結合させる反応も触媒する。2-ketobutyrate の構造はピルビン酸に非常に良く似ているので、下の反応式を見れば同じ酵素で触媒されることが理解できるだろう。この反応はイソロイシン生合成の第一段階である。

上記の二つの反応はいずれも不可逆であり、Val と Ile の生合成経路はここから完全に分岐する (7I)。
ALS は
発酵における機能
いくつかのバクテリアでは、ピルビン酸から diacetyl/butanediol を生成することで発酵 fermentation にも寄与している (5I)。
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
アイソフォーム
バクテリアには複数の ALS が存在するが、単に ALS と言った場合には anabolic ALS を意味することが多い。多くのバクテリアは ALS I, II, III をもっており、kinetic properties や基質特異性が異なる (2)。I と II が系統的に近い。一種類しか ALS をもっていない種もあり、その場合は ALS III に近い。
Anabolic ALS の最適 pH は、一般に 8 前後である (9I)。
ALS |
ALS I | |
| ALS II | ||
| ALS III | ||
Catabolic ALS |
Anabolic ALS よりも最適 pH が低く、pH 6 前後が最適である (9I)。また FAD-independent である。 |
Catabolic ALS (cALS)
- 同じ反応を触媒する。
- 通常の
anabolic AHAS よりも最適 pH が低く、FAD 依存的である。 - Butanediol operon 内に遺伝子がみられ、regulatory subunit をもたない。
- Anabolic ALS との同一率 (塩基? アミノ酸?) は 30% 以下である。
ALS-like protein
ALS は動物には存在しないと考えられてきたが、ヒトで類似の構造をもつタンパク質が報告されている (5I)。
- この遺伝子は、最初は Joutel et al. 1996 によって報告された。
- いくつかの EST に含まれており、実際に発現していると考えられる。
- 大腸菌発現および in vivo complementation test で活性を検出できなかった。
- Folding, サブユニットの存在などが原因として考えられる。
- 植物の ALS は葉緑体に局在する。
- ミトコンドリア or 葉緑体移行シグナルをもっている。
構造
真核生物の ALS は、
サブユニット構造
- ただし、酵母および植物の ALS は、small subunit がなくても活性をもつ。
重要なアミノ酸残基
| アミノ酸 (生物種) | 機能 | 文献 |
|---|---|---|
Glu85 (Mycobacterium tuberculosis 他) |
ThDP との結合に関わるアミノ酸。ALS だけでなく、ThDP-dependent enzymes でよく保存されている。
|
4R |
His84, Gln86 (Mycobacterium tuberculosis 他) |
Glu85 の側鎖が正しい高次構造をとるサポートをする。 |
4R |
広告
酵素としての特徴
Thiamin diphosphate (ThDP), FAD および 2 価の金属イオンが活性の発現に必要である (2)。金属イオンは、ThDP が酵素に結合するために必要 (4I)。
- 最初に pyruvate と酵素の複合体が形成される。この結合サイトには、2-ketoburyrate は結合しにくい。
- 2 段階目で、複数の 2-keto acids がこの複合体に結合しようとする。
- Natural substrate でない 2-keto acid の 2-ketovalerate は、ピルビン酸の 2.5 倍の親和性をもつ。
- しかし、ピルビン酸と 2-ketoburyrate に対する特異性は、他の 2-keto acids によって左右されない。
- ピルビン酸と 2-ketoburyrate に対する特異性は、pH や feedback inhibition などの影響を受けない。
- これらは、大腸菌の 3 種の ALS に共通した特徴である。
- AHAS I は特に2-ketoburyrate に対する特異性が低かった。
Regulatory subunit と活性
ALS には regulatory および catalytic subunit があり、ヘテロダイマーは catalytic 単体の 5 倍以上活性が高い (3)。Catalytic サブユニットだけでも active である (4I) という報告もあり、regulatory subunit は補助的な機能をもつと考えられる。
BCAA によるフィードバック阻害
ALS には、一連の酵素反応の最終産物である
- 実際に、Arabidopsis の種から精製した ALS ではフィードバック阻害がある。
- しかし、大腸菌で発現させた Arabidopsis ALS にはフィードバック阻害なし。
- おそらく、大腸菌の regulatory subunit が植物 ALS とはうまく働かないのだろう。
- ただし、folding など実験系がうまく動いていない可能性も否定はできない。
広告
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
References
- 岩波理化学辞典 第 4 版.
|
このサイトでは、私が持っている 1987 年の第 4 版を引用していることが多い。1998 年に第 5 版が発行されている。 ネット情報の問題点の一つは、信頼できる定義になかなか出会えないことである。Wikipedia には定義らしいことが書いてあり、普段の調べ物には十分なことも多いが、正式な資料を作るときにはその引用は避けたいものである。 そんなときに役に立つのが理化学辞典や生化学辞典。中古でも古い版でもよいので、とにかく 1 冊持っておくと仕事がはかどる。 |
Gedi and Yoon 2012a (Review). Bacterial acetohydroxyacid synthase and its inhibitors – a summary of their structure, biological activity and current status. FEBS J, 279, 946-963.Binder 2010 (Review). Branched-chain amino acid metabolism in Arabidopsis thaliana. The Arabidopsis Book.Baig et al. 2014a. Structural and functional significance of the highly-conserved residues in Mycobacterium tuberculosis acetohydroxyacid synthase. Enz Microbiol Tech 58-59, 52-59.Duggleby et al. 2000a. Expression in Escherichia coli of a putative human acetohydroxyacid synthase. J Biochem Mol Biol 33, 195-201.Smith et al. 1989a. Functional expression of plant acetolactate synthase genes in Escherichia coli. PNAS 86, 4179-4183.Gollop et al. 1989a. Kinetics and Mechanism of Acetohydroxy Acid Synthase Isozyme III from Escherichia coli. Biochemistry 28, 6310-6317.Singh et al. 199sa. Arabidopsis acetohydroxyacid synthase expressed in Escherichia coli is insensitive to the feedback inhibitions. Plant Physiol 99, 812-816.Cavin et al. 1999a. Cloning of branched chain amino acid biosynthesis genes and assays of α-acetolactate synthase activities in Leuconostoc mesenteroides subsp. cremoris. Res Microbiol 150, 189-198.
