コドン: RNA 配列とアミノ酸を対応づけるルール
別ページ
広告
概要: コドンとは
mRNA は、以下の規則に従って アミノ酸 に翻訳される (2)。
- 3 つの塩基が 1 つのアミノ酸をコードする。塩基は AUGC の 4 種類であり、可能な組み合わせは 4 x 4 x 4 = 64 通りである。しかし、タンパク質を構成するアミノ酸は 20 種類なので、複数の組み合わせが同じアミノ酸をコードすることになる。これを、コドンが
縮重 degenerate しているという。とくに 3 番目の塩基が縮重していることが多い。 - コードは原則として重ならない。たとえば ATTCAAGTA という配列があり、最初のコードが ATT であった場合、次の読み枠は CAA、その次は GTA である。3 塩基ずれるということ。ただし、バクテリアや ミトコンドリア DNA では頻繁に重複がある。
- 句読点のような塩基はない。開始コドン、終止コドンが句読点にあたるとも言えるが。
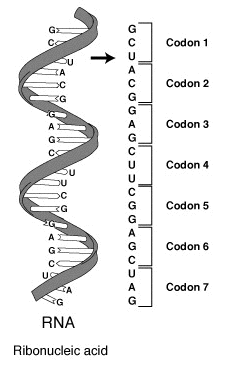
下の表は真核生物のコドン表である (上の図とともに、いずれも Public domain) 。原核生物やミトコンドリアでは違った暗号が使われるなど、現在ではさまざまなコドン表が作られている。それらは NCBI のサイト でまとめられている。
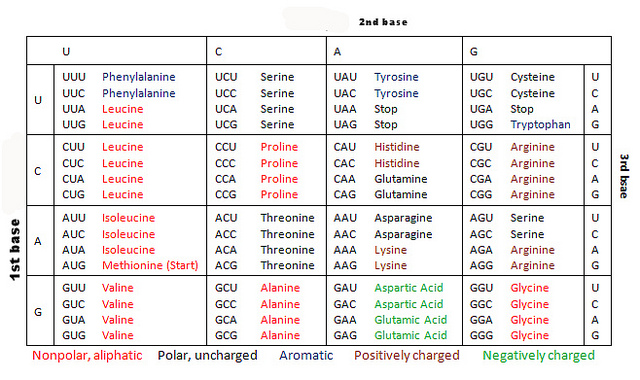
例えば、以下はバクテリア、アーキア、および植物の葉緑体で使われているコードである。
| TTT F Phe | TCT S Ser | TAT Y Tyr | TGT C Cys |
| TTC F Phe | TCC S Ser | TAC Y Tyr | TGC C Cys |
| TTA L Leu | TCA S Ser | TAA * Ter | TGA * Ter |
| TTG L Leu i | TCG S Ser | TAG * Ter | TGG W Trp |
| CTT L Leu | CCT P Pro | CAT H His | CGT R Arg |
| CTC L Leu | CCC P Pro | CAC H His | CGC R Arg |
| CTA L Leu | CCA P Pro | CAA Q Gln | CGA R Arg |
| CTG L Leu i | CCG P Pro | CAG Q Gln | CGG R Arg |
| ATT I Ile i | ACT T Thr | AAT N Asn | AGT S Ser |
| ATC I Ile i | ACC T Thr | AAC N Asn | AGC S Ser |
| ATA I Ile i | ACA T Thr | AAA K Lys | AGA R Arg |
| ATG M Met i | ACG T Thr | AAG K Lys | AGG R Arg |
| GTT V Val | GCT A Ala | GAT D Asp | GGT G Gly |
| GTC V Val | GCC A Ala | GAC D Asp | GGC G Gly |
| GTA V Val | GCA A Ala | GAA E Glu | GGA G Gly |
| GTG V Val i | GCG A Ala | GAG E Glu | GGG G Gly |
なぜコドンは縮重しているのか?
コドンの 3 番目の塩基は tRNA の 1 番目の塩基とペアを形成する。この部位では、
また、コドンが縮重していることにより、突然変異の影響を少なくすることができることも一般的なメリットであると考えられる。
同義置換と非同義置換
同じアミノ酸をコードするコドンを
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
開始コドン
開始コドンは常にメチオニン methionine をコードする。このコドンが mRNA 中で発見されるメカニズムは、翻訳の読み枠を決める上で非常に重要である。以下のように、真核生物と原核生物で大きな違いがある。
真核生物
真核生物 eukaryote の場合は、1 つの mRNA が 1 つの遺伝子をコードしている (モノシストロン性) ので、翻訳の開始は比較的単純である。すなわち、
原核生物
原核生物 prokaryote では、mRNA が複数の遺伝子を含む (ポリシストロン性)。したがって、翻訳の開始点となる AUG が 1 つの mRNA 中に複数存在する。
原核生物の mRNA では Shine-Dalgarno 配列 が開始メチオニンの上流に存在し、これが翻訳開始の合図になる。
タンパク質の N 末端に存在するメチオニンは、ホルミル基 (formyl group; 官能基の一覧) で修飾された formylmethionine (fMet) である (2)。
終止コドン
終止コドンには tRNA でなく
レアコドンとは
コドンの縮重のため、一つのアミノ酸に対して複数のコドンが存在する場合がある。たとえば、ロイシンは 4 種類のコドンにコードされる。
このような場合、
タンパク質を発現させる際に、宿主として用いる大腸菌や酵母におけるレアコドンが発現配列中に含まれていると、収率が低下する可能性がある。これは、以下のいずれか (または両方) の方法で対処する。
- ベクターの配列を変更し、レアコドンを含まないようにする (手間がかかる)。
- 宿主内で、発現量が少ない tRNA を補充する。つまり、レアコドンの tRNA がコードされているような発現ベクターを使う。
ATGme では、DNA シークエンスとコドン表を submit することで、コドンの使用頻度を調べることができる。コドン表は、上記の Codon Usage Database のものを使用する。
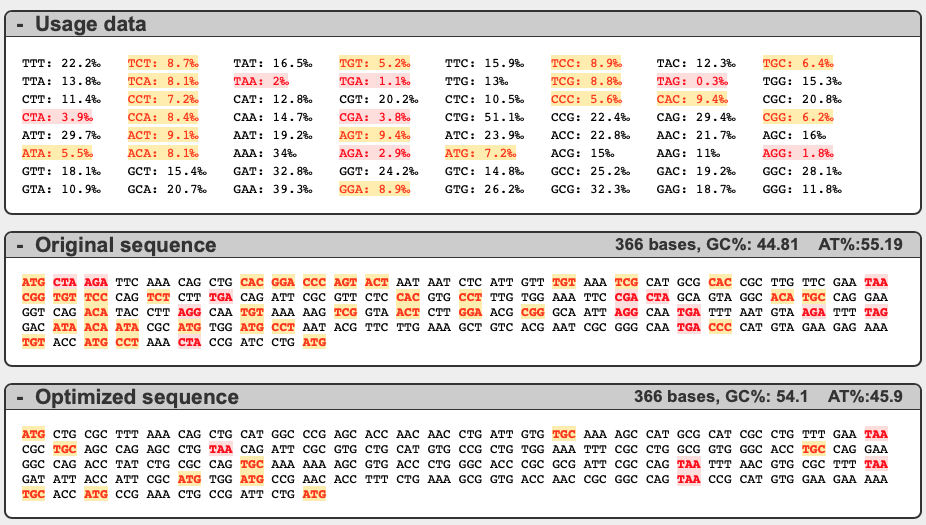
これが解析結果。元の配列に含まれているコドンのうち、レアコドンが赤で、やや使用頻度の低いコドンが黄色で表示される。Optimized sequence として、アミノ酸配列を変えずにレアコドンを除いた配列も示してくれる。
広告
References
- pETシステムにおけるタンパク質発現誘導のポイント. Link: Last access 2021/03/20.
- Amazon link: ストライヤー生化学
: 使っているのは英語の 6 版ですが、日本語の 7 版を紹介しています。参考書のページ にレビューがあります。
- Amazon link: Pierce 2016. Genetics: A Conceptual Approach
: 使っているのは 5 版ですが、6 版を紹介しています。
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
