リアルタイムPCR: 原理、プロトコール、データ解析など
UBC/experiments/rna/real-time
このページの最終更新日: 2025/11/23関連ページ
広告
原理と概要
リアルタイム PCR とは、PCR を使って DNA を定量する実験法である。「定量的リアルタイム PCR」とほぼ同じ意味で使われる。
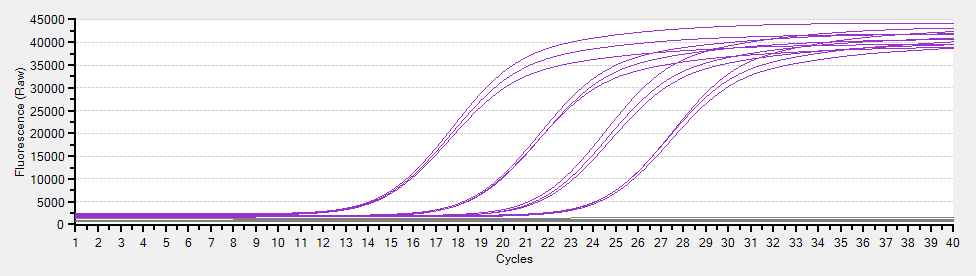
PCR 溶液中に DNA を蛍光検出する物質を入れ、それを用いて一サイクルごとに DNA 量を測定する。通常の PCR では、反応が終了して アガロースゲル電気泳動 などを行うまで、増幅された DNA は確認できない。しかし、リアルタイム PCR では、サンプルごとに以下のような DNA 増幅パターンを得ることができる。
定量の原理は、「標的 DNA を一定の量まで増幅するのに何サイクル必要だったか」である。非常に単純化した例を挙げれば、PCR をしていないサンプル A と、1 サイクルだけ PCR したサンプル B の DNA 量が同じだったとする。この場合、B では DNA が PCR によって 2 倍に増えてようやく A と同じになったと考える。したがって、A にはもともと B の 2 倍量の DNA が含まれていたことになる。
この「標的 DNA を一定の量まで増やすのに必要なサイクル数」を cycle threshold 値 (Ct 値) という。リアルタイム PCR で非常に重要な概念である。このページでは、リアルタイム PCR に関する理論的な背景を中心にまとめている。
絶対定量と相対定量
リアルタイム PCR には、DNA の絶対量を算出する
絶対定量では、濃度がわかっている DNA または RNA についてリアルタイム PCR を行い、その Ct 値を使ってサンプルに含まれる DNA 量を計算する。
相対定量は、一般に mRNA から逆転写した cDNA を鋳型として、興味のある遺伝子 (標的遺伝子 target gene) を定量する遺伝子発現解析に使われる。相対定量において標準化に使われる遺伝子を
遺伝子発現解析をする場合、絶対定量と相対定量には以下のような違いがある。
- 最初の組織量や RNA の抽出量が異なると、絶対定量では mRNA 量が大きくばらついてしまう。また、脂肪組織などそもそも mRNA の総量が違う組織での議論が難しい。
- 相対定量ではこの心配はないが、内部標準遺伝子の mRNA 量が本当に均一であるのかという問題が生じる。
どちらが優れた方法というわけではなく、そのデータを使って何を言いたいのかをよく考え、定量法を決定すべきである。
増幅産物の検出: SYBR か TaqMan probe か
リアルタイム PCR 中に DNA の濃度を測定する方法は、大きく分けて SYBR と TaqMan probe の 2 種類がある。
Ct 値の算出方法
Ct 値は、かつては増幅が指数関数的に増幅している区間で任意に定められる threshold と、増幅曲線の交点と定義されていた。このようにして Ct 値を定める方法を Crossing point 法という。
新しい方法として、増幅曲線の 2 次導関数が最大となる点を Ct 値とする 2nd derivative maximum 法がある。これはつまり、もっとも増幅速度の変化率が大きい時点を Ct とする方法で、threshold 値を設定せずにすみ再現性が高いという利点がある。しかし、2024 年現在では、まだ一部の解析ソフトにしかこの機能は実装されていない。
プライマーの設計
まず、以下がプライマー設計の際の一般的な注意点である (5)。
- プライマーは 17 - 25 塩基。
- GC 含量 40 - 60% 程度。T および C の連続、A および G の連続配列も避ける。
- Forward, reversea primers の Tm 値を揃える。
- BLAST で配列を確認する。普通の BLAST ではなく Primer-BLAST tool という専用のページが NCBI にある。
- 3' 末端の塩基、T はダメ、G または C が望ましい。ただしこれには諸説あり、A や T でも問題ない場合も多い。
- cDNA を使って発現量を調べるときは、可能ならばイントロンを挟むように設計する。これによってコンタミしたゲノム DNA からの増幅を避けることができる。
リアルタイム PCR プライマーでは、とくに SYBR での定量の場合は非特異的産物が許されないなど厳しい条件がつくので、さらに以下のような点を考慮する必要がある。
> リアルタイム PCR プライマー設計に関するタカラバイオのガイドライン (5)。
- 増幅産物が短い (80 - 150 bp 程度、300 bp ぐらいまでは許容範囲)。
- 標的 DNA 配列に安定してアニーリングできる。つまり Tm が適切。私は 60°C にセットしている。
- PCR の反応効率が良い。プライマーダイマーなどができにくい。
- 特異性が高い。非特異的な増幅産物ができない。
- 増幅効率が 100% に近い。上記の諸条件を満たした結果として達成される。
増幅効率の計算
PCR は原理的には 1 サイクルで DNA を 2 倍に増幅するが、実際の反応は理論通りには進まない。つまりプライマーによって、1 サイクルの増幅が 1.98 倍であったり 2.05 倍であったりする。増幅効率の差がわずかであっても、その数値はサイクル数分の累乗で効いてくるため、通常は 40 サイクル回すリアルタイム PCR では、大きな影響を及ぼすことがある。
ThermoFisher のページ に詳しい説明があるので参照のこと。
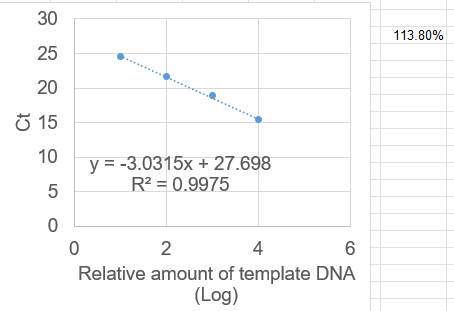
プライマーの増幅効率は、以下のような実験で算出することができる。
- 標的遺伝子を増幅可能な DNA の希釈系列を準備し、それぞれについてリアルタイム PCR をする。
- 希釈系列は、たとえば 2, 5 および 10 倍で、各 5 サンプルずつ (6)。
- 私は 10, 100, 1000, 10000, 100000 倍あたりで triplicate を使うことが多い。
- 相対 DNA 量と Ct 値をプロットし、直線で回帰する。R > 0.99 でなければならない。DNA 量は対数軸にプロットする。
- 増幅効率が 100% のとき、傾きは -3.322 になる。傾きの値と以下の式から、増幅効率を計算する。
増幅効率 = [10(-1/slope)] - 1
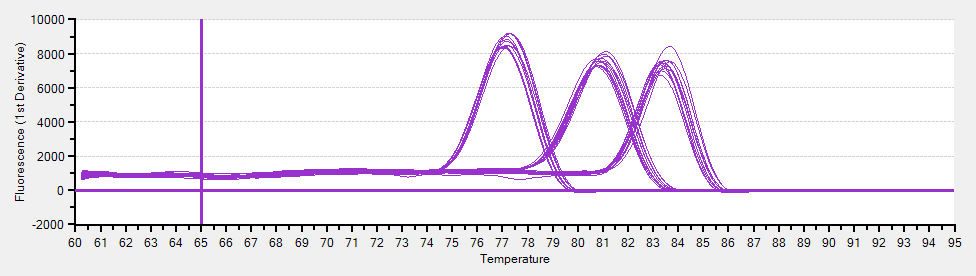
融解曲線分析
SYBR でリアルタイム PCR を行う場合には、目的の産物のみが増幅されていることを確認する必要がある。融解曲線分析 dissociation curve analysis or melting curve analysis はそのための方法で、PCR のあとに一定のプログラムを走らせることで行う。なお、増幅産物のアガロールゲル電気泳動も有効である。
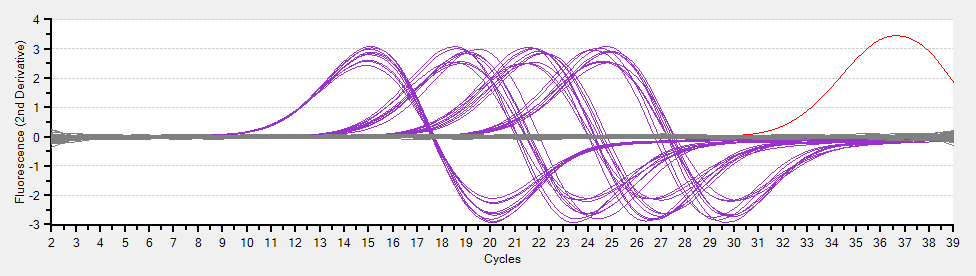
以下は 2nd derivative という図。何を表しているのか、よくわからない。調べて追記する。
その他
逆転写の効率
リアルタイム PCR で測定できるのは、サンプル内に含まれる cDNA の量である。論文では、このデータが mRNA 量を表していると考え、mRNA expression または gene expression という言葉を使って議論されることが多い。しかし、この議論をする際には以下のような点を考慮する必要がある。
- 逆転写の効率が一定とは限らない。mRNA が高次構造をとっていたら、その遺伝子だけ逆転写の効率が低いかもしれない。
- 同様に、逆転写に oligo dT primer を使っている場合、プライマーが 3' 末端から遠いと、逆転写の効率が低いかもしれない。
以上のような問題は、random primer と oligo dT primer の混合物を使って逆転写することで、かなりの部分解決することができる。
cDNA は希釈すべきか
ThermoFisher の Youtube がある。
- PCR 阻害物質 の持ち込みを気にしているなら、問題にならないことが多い。逆転写の阻害の方が起こりやすい。cDNA を希釈するよりは、RNA をきれいにしておくべき。
- ただし、逆転写 buffer の多量の持ち込みはリアルタイム PCR を阻害する。リアルタイム mixture の 20% 以上 cDNA を入れないこと (ただし、20% はかなりの量)。
- 18S のような高発現の内部標準遺伝子の立ち上がりが早すぎて、threshold line がうまく引けなくなることがある。これは希釈するよい理由だが、ターゲット遺伝子の Ct が大きくなりすぎないように注意する必要がある。
広告
References
Nakagawa et al. 2009a. Sweet taste receptor expressed in pancreatic b-cells activates the calcium and cyclic AMP signaling systems and stimulates insulin secretion. PLoS ONE 4, e5106.Bartelt et al. 2011a. Brown adipose tissue activity controls triglyceride clearance. Nat Med 2, 200-206.Livak and Schmittgen 2001a. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ddct method. Methods 25, 402-408.Schmittgen and Livak 2008a. Analyzing real-time PCR data by the comparative CT method. Nat Protoc 3, 1101-1108.- タカラバイオ, リアルタイム PCR 実践編 - プライマー設計ガイドライン.Link: Last access 2018/06/25.
- Qiagen: How do I determine the amplification efficiency of my qPCR assay? Link: Last access 2018/07/03.
Nakagawa et al. (2009a) is an open-access article distributed under the terms of the Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction in any medium, provided the original author and source are credited. Also see 学術雑誌の著作権に対する姿勢.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
