Shine-Dalgarno 配列:
原核生物 RNA のリボソーム結合部位
広告
概要: Shine-Dalgarno 配列とは
Shine-Dalgarno 配列とは、原核生物 prokaryotes の mRNA で開始コドン AUG の上流に存在する共通配列である。UAGGAGGU のように、プリン塩基に富んだ 3 - 9 塩基の配列である (1)。AGGAGG とされることもある。
Shine-Dalgarno の配列は 16S rRNA の配列と相補的であり、ゆえに
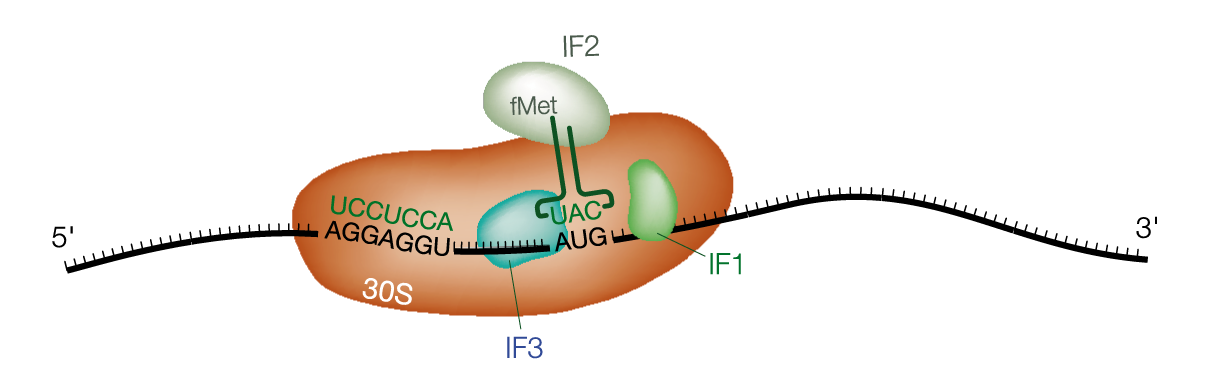
真核生物の mRNA は、1 つの mRNA が一つの タンパク質 をコードする
mRNA の 5' 側に付加される Cap 構造がリボソームの結合部位である (→ mRNA のプロセシング)。
これに対して、原核生物の mRNA は、
- 5' Cap 構造がない
- 1 つの mRNA が複数のタンパク質をコードする
ポリシストロン性 である
という特徴をもっている。翻訳は 5' 側から始まり、最初のタンパク質の終止コドンに達した時点でリボソームが遊離するため、mRNA 中に複数のリボソーム結合部位がなければならない。
Cap 構造のような化学構造ではなく、rRNA と相補的な RNA 配列を使ってリボソームを引き寄せるのは合理的な方法と言える。
真核生物にも Kozak 配列という類似の配列がある。これは AUG を含む配列で、翻訳の効率を上げる (1)。
発現ベクターを作る際に、Shine-Dalgarno 配列を加える必要はある?
タンパク質の大腸菌発現は、分子生物学でよく行われる基本的な実験の一つである。このサイトには、以下のような関連ページがある。
原核生物である大腸菌で発現させるため、発現ベクターには Shine-Dalgarno 配列が必要である。ベクターをデザインするときに、この配列を加える必要があるのだろうか?
これはよくある疑問で、ネット上でいろいろ情報がみつかる。基本的には、通常の発現ベクターならリボソーム結合配列がすでに含まれているので、ベクター構築の際に考慮する必要はないというのが回答である。
- ThermoFisher FAQ: 目的遺伝子をクローニングするとき、リボソーム結合部位(RBS)や Kozak 配列を含める必要がありますか? 発現ベクターにはリボソーム結合配列や転写開始部位が含まれているので、cDNA の配列をそのまま使えばよいという回答。
オペロンと ribosome recycling
原核生物の一種である 大腸菌 では、遺伝子の約 25% がオペロンを形成している (3)。オペロンでは、しばしば上流の遺伝子のストップコドンと、下流の遺伝子のスタートコドンが塩基を共有している。たとえば、TAA と ATG が重なって TAATG となり、共有されている A が 2 回読まれるということがあるらしい。
この場合、翻訳はどのように行われるのだろうか? いくつか異なるメカニズムがあるようだ。
まずは ribosome recycling という現象 (参考)。最初の遺伝子から合成されたポリペプチドが離れても、ribosome が DNA に結合したままとなり、そのまま翻訳が続く。
また、上流の遺伝子の一部が Shine-Dalgarno 配列になっているパターンもある。
直接は関係ない話だが、mRNA に終止コドンが存在しない場合、リボソームは mRNA の終端でペプチジル tRNA などとともに nonproductive translation complex NTC という複合体を作って立ち往生してしまうらしい。これを解放する機構というのもあるようで、興味深い。
References
- Amazon link: Pierce 2016. Genetics: A Conceptual Approach
: 使っているのは 5 版ですが、6 版を紹介しています。
- By <a href="//commons.wikimedia.org/wiki/User:Fdardel" title="User:Fdardel">Fdardel</a> - <span class="int-own-work" lang="en">Own work</span>, CC BY-SA 4.0, Link.
Salgado et al., 2000a. Operons inEscherichia coli: Genomic analyses andpredictions. PNAS 97, 6652-6657, 2000.阿保、茶谷 2015a. 大腸菌の持つ二段構えのリボソーム解放機構. 生化学 87, 736-740.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
