制限酵素によるベクター構築:
プロトコール、プライマー設計の方法など
UBC/experiments/dna/vector_restriction_enzyme
このページの最終更新日: 2025/11/23- 概要
- PCR
- 制限酵素処理
ライゲーション以降は別のページに。
- ライゲーション
- シークエンス
広告
概要
この作業は、一般に以下のような流れで行われる。問題がなければ 1 週間程度で完了する実験である。
- 制限酵素サイトを含むプライマーで目的遺伝子を増幅する。
- 制限酵素の選択
- プライマーの設計
- PCR
- PCR 増幅産物、発現ベクターを制限酵素で処理する。
- 両者を混合し、ライゲーション。大腸菌を使って完成したプラスミドを増幅。
- シークエンシングによって配列を確認。
1. 制限酵素サイトを含むプライマーで目的遺伝子を増幅する
1-1. 制限酵素の選択
制限酵素は、以下の条件を満たすものが理想である。ただし、後述するように対処法もある。
- ベクターのマルチクローニングサイト中に含まれる。
- ベクターのその他の部位、組み込む DNA 配列中には含まれない。
- 付着末端 cohesive end (突出末端 protruding end ともいう) で DNA を切断するため、組み込みの方向を決めることができる。
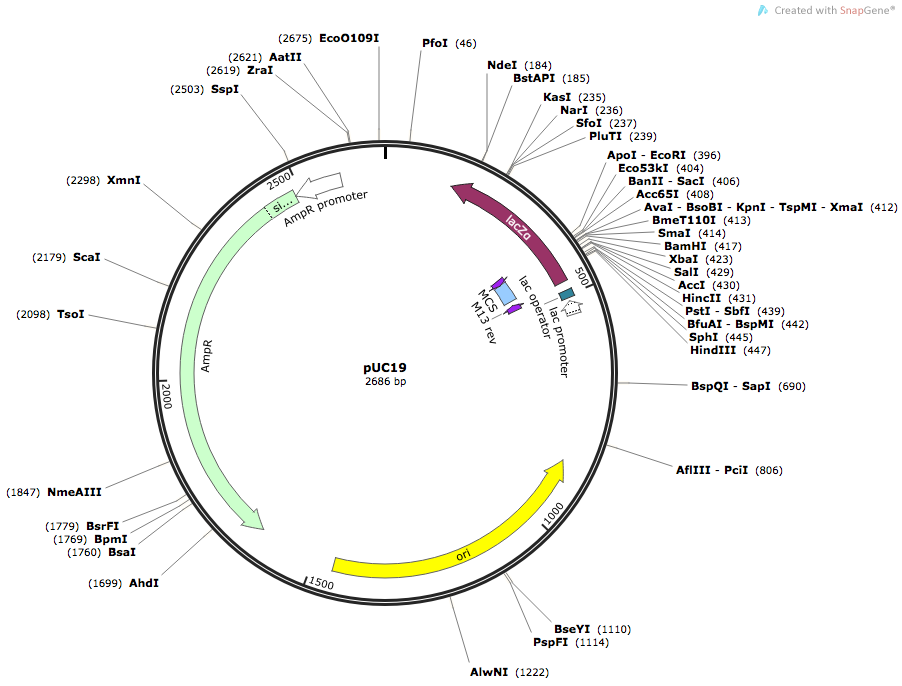
たとえば、マウス mouse の メラノコルチン-3受容体 遺伝子を上記の pUC19 ベクターに組み込みたいとしよう。PubMed で検索対象を Nucleotide にし、mouse melanocortin で検索すると、melanocortin 3 receptor (Mc3r), mRNA (NM_008561.3) がみつかる。翻訳領域の塩基配列は以下の通り。
制限酵素サイトを調べる方法はいろいろあるが、Enzyme X が無料で使い易いのでお勧め。
マルチクローニングサイト中にある、一般的な制限酵素 BamHI、XbaI、SalI、HindIII などの認識配列は全て遺伝子中にみつかってしまった。珍しい組み合わせだが、5' 側は AvaI、3' 側は SphI という選択肢しかなさそうなので、これらの制限酵素を使うことにする。
良い制限酵素が見つからない場合は、次のようなオプションがある。
- 付着末端酵素を使って fill in する。
- 遺伝子を 1 箇所切ってしまう酵素を使って、2 段階でベクター構築する。
- In-fusion など他の方法を使う。
1-2. 制限酵素サイトのついたプライマーの設計
制限酵素サイトを付加したプライマーを用いる。設計方法は、制限酵素付きプライマーの設計 を参照のこと。上記の配列の翻訳領域のみをベクターに組み込みたいとして、Forward primer は「ATG から始まる 20 塩基程度の配列 (太字)」 + 「制限酵素の認識配列 (下線)」 + 「5 - 6 塩基のスペーサー配列」である。
Reverse primer は「ストップコドンを含む 20 塩基程度の配列 (太字)」 + 「制限酵素の認識配列 (下線)」 + 「5 - 6 塩基のスペーサー配列」である。
Forward primer:
Reverse primer:
この配列をもとに、Tm 値、二次構造の有無などの基本的な点も考慮して最終的な配列を決定する。
1-3. PCR
この PCR は、プライマーに余計な配列がついている分、通常の PCR よりも難しいかもしれない。うまく増幅されないときは、以下のような方法で改善する場合がある。
- 一度、余計な配列を含まないプライマーで PCR をしてから、その増幅産物またはそれを組み込んだクローニングベクターを鋳型に PCR する。
このほか、通常の PCR の Tips ももちろん有効である。
制限酵素処理
PCR 産物
PCR 反応液の一部を電気泳動 electrophoresis に供して増幅を確認したら、残りを制限酵素処理に用いる。
典型的な反応系は次の通り。37℃ で 1 時間。
| PCR 産物 | |
| 10 x 緩衝液 | |
| 制限酵素 restriction enzyme | 1 µl |
| 滅菌水 sterile water | 全量 10 µl にする |
注意点
- 制限酵素の量は全体の 10% を超えないように。
広告
References
- Snap Gene pUC 19 vector. Link.
- 生体機能物質工学実験 II. 2-4. PCR 産物とプラスミドベクターの制限酵素処理. Link.
- Clontech, Takara Q & A. ライゲーション一般. Link.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
