Gene Ontology (GO): 定義、使い方、問題点など
- 概要: Gene ontology とは
- GO の問題点
広告
概要: Gene ontology とは
遺伝子の配列や名前を一次情報とすると、付加情報はそれらを整理するための高次元の情報、すなわち
GO は、以下のように 3 つに分類されており、それぞれが階層構造をもっている。
Molecular Function |
翻訳産物の機能。e.g. binding, electron career activity. |
Cellular Component |
翻訳産物の細胞内局在。e.g. mitochondria. |
Biological Process |
翻訳産物が関わる生物学的なプロセス。e.g. lipid metabolism, amino acid metabolism. |
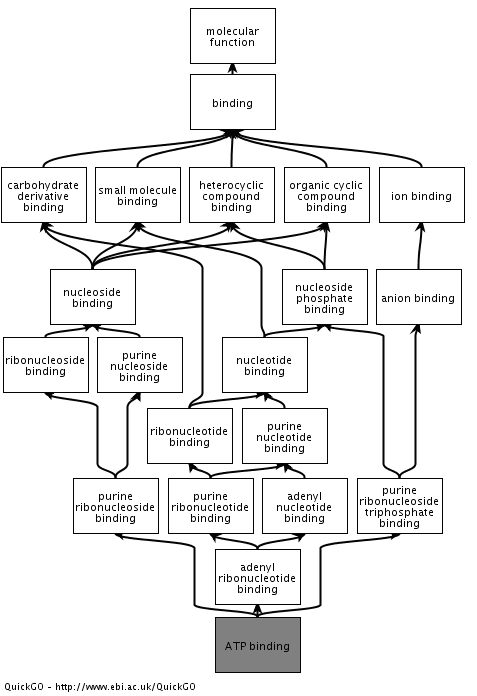
たとえば、ATP と結合する分子には ATP binding という Molecular Function の GO が振られているが、これには図のような上位概念がある。
したがって、ATP binding という GO をもつ分子は、自動的に binding という GO ももっていることになる。
> GO には以下のような特徴がある (1)。
- GO をもっていない遺伝子もある。とくに、遺伝子の機能解析が進んでいない生物。
- 1 つの遺伝子がもっている GO は、1 つだけとは限らない。
- 階層構造をもっているため、1 つの GO にはたくさんの上位 GO が付属している。
GO の問題点
かなり個人的な話になるが、私は昔からこの GO というのが気に入らなかった。
この言葉は、マイクロアレイとか NGS などの大規模解析が発展してきた 2000 年代初頭からよく目にするようになる。そもそも遺伝子の機能は一つの生物でも非常に多様であり、ましてや昆虫などの非モデル生物のホモログでは、ヒトの遺伝子と違う役割を持っている場合が容易に想像できる。
そのような多彩な機能を適当なカテゴリーにあてはめて、なんか「わかった気になる」解析方法だなあという気がしていた。EST の結果を GO で色分けした円グラフみたいのが一時期よくあったけど、あれで何が言えるのか、未だによくわからない。
さらに個人的なことになるが、私の GO 嫌いを加速させた出来事がある。あるとき、自分の論文で納得いかないながらも空気を読んで GO を使ったことがある。その際の査読者のコメントが、「GO や KEGG みたいなキュレーションベースの方法はもう古い。科学者は GSEA などの発現量ベースの方法にシフトしている」。
流行りに背を向けて我が道を行くことこそ科学者の本懐、という信念を曲げて、日和って GO を論文に使った自分を恥じたエピソードである。
GSEA 解析
GO ではないが、上のエピソードに関連して GSEA について簡単にまとめておく。内容次第で別のページを作る。
広告
References
- GO 解析 (知識) Link: Last access 2020/06/08.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
